Le Néolithique s'est diffusé en Europe à partir du Proche-Orient en suivant deux routes majeures: la route continentale en remontant la vallée du Danube associée à la culture LBK en Europe Centrale, et la route Méditerranéenne associée aux cultures Impressa et cardiale dans le sud de l'Europe. Les études génétiques précédentes sur des squelettes du Néolithique ont montré:
- un remplacement de la diversité génétique mitochondriale avec l'arrivée des fermiers du Proche-Orient
- un pool de gènes différent associé au groupe des agriculteurs issus des vagues continentale et Méditerranéenne
S'il y a eu une claire discontinuité génétique mitochondriale entre les chasseurs-cueilleurs et les premiers fermiers en Europe Centrale, on remarque une assimilation croissante des lignées maternelles de chasseurs-cueilleurs dans le pool génétique des fermiers du Néolithique moyen et récent.
Les deux vagues principales de Néolithisation sont entrées en contact sur un territoire qui s'étend en France. D'un côté, l'archéologie indique une nette influence culturelle de la culture rubanée dans le nord-est de la France, en particulier le bassin Parisien. D'un autre côté, l'archéologie observe une influence Méditerranéenne dans les cultures du bassin Parisien. L'analyse de l'ADN ancien peut permettre de mettre en évidence la rencontre des deux vagues Néolithiques dans le centre de la France. Malheureusement, actuellement seules trois séquences mitochondriales sont disponibles sur des squelettes du Néolithique Moyen de l'Ouest de la France, et aucune séquence du Néolithique Ancien n'est disponible en France.
Maïté Rivollat vient de publier un papier intitulé: When the Waves of European Neolithization Met: First Paleogenetic Evidence from Early Farmers in the Southern Paris Basin dans lequel elle étudie l'ADN ancien de squelettes issus de la nécropole de Gurgy située sur les rives de la rivière Yonne. Ce site est daté entre 5000 et 4000 av. JC. avec une occupation principale entre 4900 et 4500 av. JC. correspondant à la transition entre le Néolithique Ancien et le Néolithique Moyen en France.
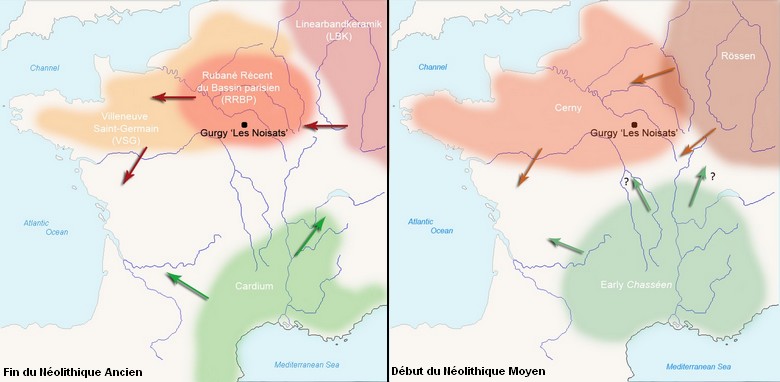
Gurgy est une nécropole sans monument funéraire apparent contrairement aux autres nécropoles de la région. Elle regroupe la sépulture de 128 individus. Les pratiques funéraires se rapprochent de celles qui existent dans l'ouest de la Suisse. L'homogénéité des sépultures fait penser à une unique population appartenant à une seule culture archéologique. Les auteurs ont analysé l'ADN de 102 individus en testant l'ADN mitochondrial et du chromosome Y à partir d'un ensemble de 38 SNPs représentatifs des principaux haplogroupes européens (28 SNPs mitochondriaux et 10 SNPs du chromosome Y). De plus la région HVR1 de l'ADN mitochondrial a été séquencée.
L'haplogroupe mitochondrial de 55 individus a été déterminé: H (36%), K (20%), U (16%), J (11%), N1a (5,5%), T (3,6%), X (3,6%), V (1,8%) et HV (1,8%). Les séquences HVR1 ont pu être déterminées pour 39 échantillons. 27 haplotypes différents ont été obtenus. Malheureusement aucun résultat n'a été obtenu sur le chromosome Y. 5 haplotypes sont partagés par au moins 2 individus laissant supposer des liens maternels entre ces échantillons. Notamment 3 individus de l'haplogroupe K ont le même haplotype et sont enterrés proche l'un de l'autre.
Les auteurs ont ensuite comparé ces résultats avec ceux d'études précédentes. Ainsi la diversité génétique de la nécropole de Gurgy est similaire à la diversité génétique des groupes Néolithiques d'Europe Centrale, mais est supérieure à la diversité génétique obtenue dans la péninsule Ibérique. 4 haplotypes de Gurgy sont communs avec des haplotypes de nombreux autres sites préhistoriques. 3 sont communs avec des haplotypes de seulement quelques sites et enfin 20 haplotypes sont spécifiques à Gurgy. Un réseau a été construit pour comparer ces différents haplotypes:
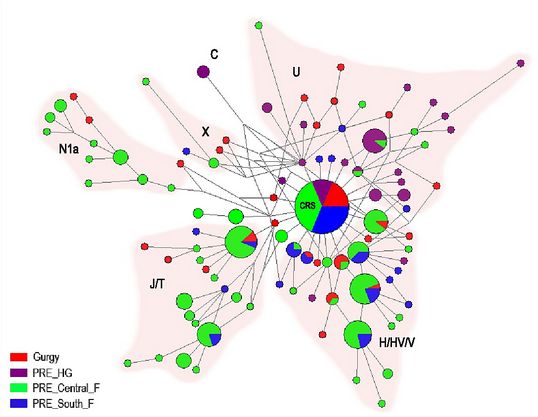
Dans la figure ci-dessus, PRE_HG correspond aux échantillons chasseurs-cueilleurs plus anciens que 4000 av. JC., PRE_Central_F correspond aux fermiers d'Europe Centrale plus anciens que 4000 av. JC. et PRE_South_F correspond aux fermiers du Sud de l'Europe plus anciens que 4000 av. JC. Cette figure n'est pas complètement satisfaisante car elle visualise des haplotypes basés uniquement sur la région HVR1. Cependant elle montre clairement que la majorité des haplotypes de Gurgy dérive d'haplotypes des fermiers d'Europe Centrale ou du Sud.
L'Analyse en Composantes Principales montre clairement que les individus de Gurgy sont éloignés des chasseurs-cueilleurs Européens, et se trouvent entre les fermiers d'Europe Centrale et les fermiers Méditerranéens:
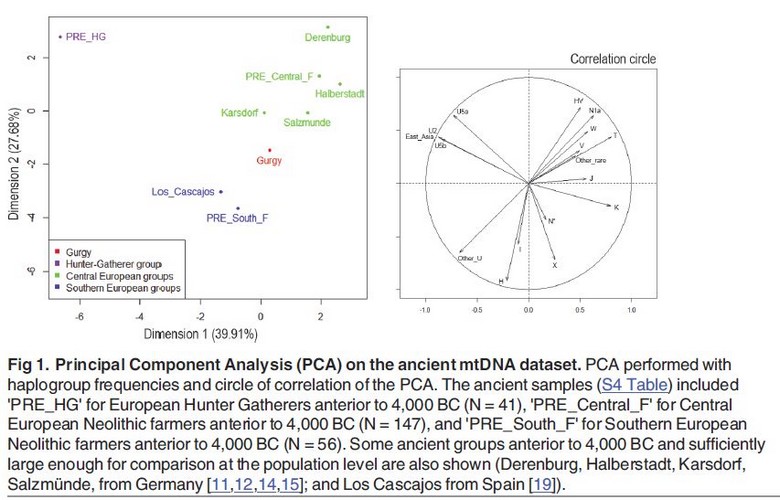
Les mesures de distance génétique confirment que le groupe de Gurgy est éloigné des groupes de chasseurs-cueilleurs, et plus proche des groupes de fermiers. Cependant il est plus proche des fermiers du Néolithique récent que des fermiers du Néolithique ancien, probablement à cause du pourcentage relativement élevé d'échantillons de l'haplogroupe U (16%) alors qu'il ne dépasse pas quelques pourcents dans le Néolithique Ancien d'Europe Centrale:

De manière intéressante, le groupe de Gurgy possède 3 haplotypes de l'haplogroupe N1a qui est considéré comme un marqueur du Néolithique Danubien. Il est donc certain que ce site archéologique a reçu un influx génétique en provenance des fermiers LBK d'Europe Centrale. Cependant comme le montre l'Analyse en Composantes Principales, ce site a probablement également reçu un influx génétique en provenance d'Europe Méditerranéenne.

