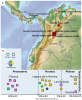
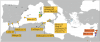
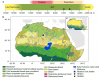
La civilisation égyptienne a développé une architecture monumentale et un système de croyances stable pendant des millénaires entre 3150 et 30 av. JC. A la suite de la réunification politique de l’Égypte à la fin du quatrième millénaire av. JC., l'Ancien Empire a connu des progrès considérables dont […]