En 2017 et 2018 un cimetière campaniforme a été découvert sur le site de Reguisheimer Feld à Ensisheim en Alsace, lors d'une fouille préventive au préalable à l'établissement d'une zone d'activité. Tout d'abord un ensemble de neuf sépultures campaniformes ont été retrouvées à l'est du site. Un peu plus tard deux tombes campaniformes isolées ont été mises au jour. Dans la figure ci-dessous, les sépultures campaniformes sont représentées en bleu foncé:

Parmi cet ensemble, six tombes ont livré du mobilier archéologiques: six gobelets campaniformes, un pichet, une céramique, deux brassards d'archer, quatre pointes de flèches, trois boutons à perforations en V, un pendentif arciforme et deux bracelets en argent:

La tombe 221 est celle d'un homme adulte en position contractée sur le côté gauche et sa tête est positionnée au nord. Cette position est typique des sépultures campaniformes. Elle est très bien dotée avec un vase campaniforme, des boutons à perforation en V, un brassard d’archer, une pointe de flèche et un pendentif arciforme:

Le gobelet est bien conservé:

et le brassard d'archer possède quatre trous de perforation:

La sépulture 236 est celle d'un adulte de sexe indéterminé en position contractée sur le côté droit et sa tête est positionnée vers l'est. Elle est accompagnée d'un gobelet campaniforme. Elle présente une originalité: le dépôt d’une crémation d’un individu immature au pied. Les ossements brûlés se trouvaient probablement dans un contenant souple (cuir, tissu):

La tombe 8182 est celle d'un homme adulte en position contractée sur le côté gauche et sa tête est positionnée au nord. Elle est également bien dotée avec trois gobelets campaniformes, un brassard d'archer, une pointe de flèche et deux bracelets d'argent. Cette tombe a été endommagée, peut-être suite à un pillage, car une bonne partie des ossements du torse a disparu:

Les trois gobelets sont magnifiques dans un style différend de celui de la tombe 221:

le brassard d'archer possède également quatre trous de perforation:

Les bracelets d'argents forment une parure exceptionnelle dans une sépulture campaniforme:

Enfin la sépulture 8347 est encore celle d'un homme adulte en position contractée sur le côté gauche et sa tête est positionnée au nord. Elle est accompagnée d'un gobelet campaniforme et d'une pointe de flèche:

Le gobelet découvert au pied de la sépulture est en morceaux:

dimanche 14 décembre 2025
Le cimetière campaniforme de Reguisheimer Feld à Ensisheim en Alsace
dimanche 14 décembre 2025. Archéologie
dimanche 26 octobre 2025
Génome d'un dénisovien de l'Altaï vieux de 200.000 ans
dimanche 26 octobre 2025. ADN ancien

Les dénisoviens sont un groupe humain éteint qui a été identifié pour la première fois en 2010 à partir du génome d'une petite phalange d'un doigt découverte dans la grotte Denisova dans les montagnes de l'Altaï situées à l'ouest de la Sibérie. Cette première analyse a montré que les dénisoviens […]
samedi 18 octobre 2025
Anciens génomes de l'est du Kazakhstan entre le début du Néolithique et la fin de l'Âge du Bronze
samedi 18 octobre 2025. ADN ancien

Les populations de l'Asie Intérieure sont remarquablement diverses bien que principalement structurées le long de trois gradients reflétant différentes zones écologiques et géographiques. Les récentes études de paléo-génomique ont permis de reconstruire les différents processus démographiques de la […]
jeudi 2 octobre 2025
Mélange génétique entre les populations associées au Gravettien d'Europe de l'Est et de l'Ouest en Europe
jeudi 2 octobre 2025. ADN ancien
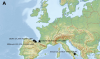
Le Gravettien est une ancienne culture paléolithique datant d'avant le dernier maximum glaciaire. Malgré une certaine similarité de la technologie, la culture Gravettienne n'était pas totalement homogène. Ainsi neuf groupes régionaux ont été identifiés archéologiquement. Les génomes Gravettiens […]
jeudi 25 septembre 2025
Une nouvelle découverte d'une sépulture campaniforme individuelle à Saint Martin la Garenne
jeudi 25 septembre 2025. Archéologie

Le musée de la préhistoire d'Île de France à Nemours présente actuellement une exposition intitulée: Pouvoir et métal - L'âge du Bronze en Île-de-France, de 2300 à 800 avant notre ère. Cette exposition démarre sur l'utilisation de l'or et du cuivre à la fin du Néolithique. A cette occasion elle […]
samedi 30 août 2025
Génomes provenant d'une communauté de la fin de l'âge du bronze enterrée dans un tumulus du nord-est de la péninsule Ibérique
samedi 30 août 2025. ADN ancien

La diversité génétique européenne a été façonnée par différents événements démographiques clés, dont la dernière transformation majeure était l'afflux de migrants issus de pasteurs de la steppe ponto-caspienne entre 3000 et 2000 av. JC. A l'âge du bronze, divers groupes culturels ont émergé et se […]
lundi 11 août 2025
Étude paléogénétique d'une nécropole de la fin de l’Antiquité et du début du Moyen Âge à Angers
lundi 11 août 2025. ADN ancien

Traversée par la Maine peu avant sa confluence avec la Loire, la ville d'Angers est occupée par l'homme depuis au moins le néolithique. À la fin de l’Antiquité, la cité des Andécaves, ancien peuple gaulois, est l’un des derniers territoires qui perdure sous l’Empire romain et reste aujourd’hui une […]
mercredi 23 juillet 2025
Histoire génomique de l'Italie du nord-est entre le mésolithique et l'âge du bronze
mercredi 23 juillet 2025. ADN ancien

Les Alpes italiennes de l'est ont joué un rôle de pont entre les populations du nord des Alpes et celles du sud des Alpes. Durant le dernier maximum glaciaire les Alpes étaient recouvertes de glaciers. Après 18.000 ans les glaciers se sont retirés peu à peu laissant la place à une population de […]
vendredi 4 juillet 2025
Génome d'un individu de l'Ancien Empire Égyptien
vendredi 4 juillet 2025. ADN ancien

La civilisation égyptienne a développé une architecture monumentale et un système de croyances stable pendant des millénaires entre 3150 et 30 av. JC. A la suite de la réunification politique de l’Égypte à la fin du quatrième millénaire av. JC., l'Ancien Empire a connu des progrès considérables dont […]
mardi 24 juin 2025
L'homme de Harbin en Chine est un Dénisovien
mardi 24 juin 2025. Protéome ancien
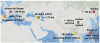
Les Dénisoviens sont un groupe d'anciens hominidés d'Asie identifié à partir de l'analyse ADN de fossiles issus de la grotte Denisova située en Sibérie. Les fossiles actuels attribués à l'homme de Denisova représentent quelques dents et quelques fragments d'os. Des analyses protéomiques précédentes […]
samedi 21 juin 2025
ADN ancien en Colombie entre 6000 et 500 ans
samedi 21 juin 2025. ADN ancien
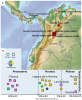
Les études précédentes de paléo-génétique ont montré que les populations Amérindiennes en dehors de la zone arctique, dérivent d'un mélange génétique entre une population est asiatique et une population sibérienne qui a eu lieu quelque part dans le nord est de l'Asie il y a plus de 20.000 ans. Il y […]
dimanche 15 juin 2025
Transition entre le Néolithique et l'Âge du Bronze à Mas d’en Boixos en Catalogne
dimanche 15 juin 2025. ADN ancien
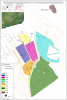
Mas d’en Boixos est le site archéologique le plus significatif de la région de Penedès en Catalogne située entre Barcelone et Tarragone. Les fouilles débutées en 1997 ont livré 450 structures datées entre le début du Néolithique et la fin de l'Âge du Fer soit entre 5500 et 200 av. JC. Ce site est […]
samedi 3 mai 2025
Continuité génétique dans la culture des Pueblos au Nouveau Mexique
samedi 3 mai 2025. ADN ancien

Les anciens pueblos créés par la culture ancestrale des Pueblos, un terme indiquant leur relation ancestrale avec les tribus amérindiennes contemporaines des Pueblos, comptent parmi les cultures archéologiques les plus reconnaissables d'Amérique du Nord. De nombreux pueblos anciens sont devenus le […]
vendredi 25 avril 2025
Anciens génomes des communautés Phéniciennes et Puniques
vendredi 25 avril 2025. ADN ancien
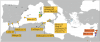
La culture phénicienne a émergé dans des cités états de l'Âge du Bronze du Levant. Au début du premier millénaire av. JC., les phéniciens ont établi un vaste réseau commercial le long des côtes méditerranéennes jusque sur les côtes sud-ouest de la péninsule Ibérique, diffusant leur culture, leur […]
lundi 14 avril 2025
La mandibule d'un homme de Denisova à Taïwan
lundi 14 avril 2025. Protéome ancien
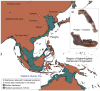
Les Dénisoviens ont été reconnus comme un groupe humain distinct des hommes modernes et des hommes de Néandertal par l'analyse ADN de restes osseux et dentaires issus de la grotte de Denisova située dans l’Altaï au sud de la Sibérie. Leur génome nucléaire a montré qu'ils formaient un groupe frère […]
lundi 7 avril 2025
Génomes anciens du Sahara vert
lundi 7 avril 2025. ADN ancien
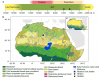
Après le dernier maximum glaciaire, une transformation climatique au Sahara a conduit à une période Africaine humide qui a été maximale entre 11.000 et 5000 ans. A cette époque le Sahara était une savane dans laquelle le paysage alternait entre couverture arborée variable, lacs permanents et système […]
dimanche 30 mars 2025
Histoire génétique de la région Rhin-Meuse
dimanche 30 mars 2025. ADN ancien
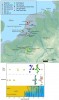
La région Rhin-Meuse s'étend au Pays-Bas, en Belgique et dans le nord-ouest de l'Allemagne. Elle est caractérisée par un environnement riche en eau. Vers 5500 av. JC. le sud de cette région voit l'arrivée de fermiers colons appartenant à la culture rubanée qui se sont installés dans des zones […]
lundi 17 mars 2025
Génomes anciens en Afrique du nord
lundi 17 mars 2025. ADN ancien

Sur la côte Méditerranéenne de l'Europe, la transition néolithique se caractérise par la migration de populations venues d'Anatolie qui ont atteint la péninsule Ibérique il y a 7500 ans. Ces populations ont absorbé jusqu'à 30% d'ascendance des chasseurs-cueilleurs de l'ouest. La culture est alors […]
mardi 4 mars 2025
Diffusion des langues celtiques
mardi 4 mars 2025. ADN ancien

Le paysage linguistique de l'Europe actuel est dominé par les langues Romanes, Germaniques et Slaves qui se sont diffusées relativement récemment durant l'Âge du Fer et après l'émergence de l'empire Romain et des migrations barbares. Cependant avant ces événements, les langues celtiques dominaient […]
mardi 25 février 2025
Anciens génomes de nourrissons sur le site de Lepenski Vir en Serbie
mardi 25 février 2025. ADN ancien

Un des rares sites archéologiques qui témoigne des interactions entre chasseurs-cueilleurs et fermiers est localisé à Lepenski Vir proche de la frontière entre la Serbie et la Roumanie. Les fouilles dans les gorges du Danube ont révélé des habitats et des sépultures fournissant plus de 500 restes […]
« billets précédents - page 1 de 33

