Qiaomei Fu vient de publier un papier intitulé: A Revised Timescale for Human Evolution Based on Ancient Mitochondrial Genomes, dans lequel il estime le taux de mutations à partir de séquences complètes de l'ADN mitochondrial issues de squelettes préhistoriques.
Les différences entre plusieurs séquences ADN correspondent aux mutations qui se sont accumulées depuis l'époque de leur ancêtre commun le plus récent (MRCA). Connaissant le nombre moyen de mutations par unité de temps, on en déduit le taux de mutations. Si on considère que ce dernier est constant au cours du temps, on peut estimer le temps de divergence entre espèces ou entre populations. Les restes fossiles ont souvent été utilisés pour estimer une date pour un MRCA de deux groupes, donnant ainsi un point de calibration pour l'estimation des taux de mutations. Cependant la rareté de ces fossiles pose problème, ainsi aucun fossile n'a été identifié pour représenter le MRCA entre humains et chimpanzés. En conséquence, les taux de mutations nucléaire et mitochondrial, pour les humains ont longtemps été débattus.
De récentes analyses des comparaisons des génomes entre parents et enfants, ont permis le calcul direct des taux de mutations par génération. De manière surprenante, ces résultats ont donné des valeurs deux fois plus faible pour le taux de mutation, que les estimations obtenues à partir des fossiles. Ceci a des impacts importants sur l'estimation de certains temps clefs dans l'évolution humaine comme la divergence entre l'homme et le chimpanzé, la divergence entre l'homme et Néandertal, ou la migration d'un groupe humain d'une région à une autre. Avec ces derniers résultats, la plupart des événements devraient être repoussée d'un facteur 2, par exemple conduisant à une estimation de la sortie de l'Afrique de l'homme entre 90.000 et 130.000 ans, ce qui est 60.000 ans plus ancien que les précédentes estimations.
Une alternative pour obtenir une meilleure précision sur l'estimation des taux de mutations, est d'utiliser des tests ADN sur des squelettes fossiles dont on connait bien la date. En comparant ce génome avec le génome d'individus contemporains, on peut mesurer le nombre de mutations et donc estimer le taux de mutations. Dans cette étude, les auteurs ont utilisé les séquences mitochondriales complètes de dix anciens hommes modernes, pour lesquels des dates radiocarbones précises sont connues. Ces dates sont comprises dans un intervalle de 40.000 ans, et sont originaires d'Europe et d'Asie de l'est.
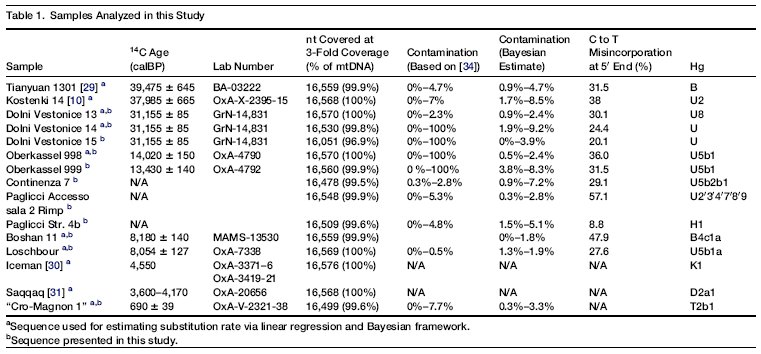
Toutes les séquences paléolithiques européennes sauf une, sont de l'haplogroupe U, confirmant ainsi les découvertes précédentes montrant la dominance de cet haplogroupe en Europe avant le néolithique. L'exception vient de l'échantillon "Cro-Magnon 1" qui est T2b1 et est daté de 30.000 ans. Comme la date estimée pour cet individu vient d'un coquillage associé, les auteurs ont redaté cet échantillon directement à partir des ossements. L'âge ainsi obtenu est seulement de 700 ans, montrant ainsi que le coquillage n'était pas réellement associé au squelette. Ce dernier a par la suite, été enlevé des collections Cro-Magnons du musée du Louvre. Un arbre phylogénétique de l'haplogroupe U, a été construit à partir des échantillons restants, et avec 63 séquences d'individus contemporains. Le résultat montre clairement que les 4 individus pré-glaciaires (en rouge) sont sur une plus courte branche que les 4 individus post-glaciaires (en bleu):

La séquence Tianyuan de l'est de la Chine se retrouve à la base de l'haplogroupe B.
La diversité des européens préhistoriques est deux fois plus faible que la diversité des européens d'aujourd'hui, mais 1,5 fois plus grande que celle des Néandertaliens.
Le taux de mutations pour l'ensemble de la molécule mitochondriale est estimé à 1,92 10-8 par site et par an, et 1,25 10-8 par site et par an pour la région codante seulement, avec un modèle de régression linéaire. Par contre pour un modèle Bayésien, l'estimation est de 2,67 10-8 par site et par an pour l'ensemble de la molécule mitochondriale et 1,57 10-8 par site et par an pour la région codante. Si maintenant on inclut les résultats des Néandertaliens dans le calcul, les estimations des taux de mutations sont proches de ceux calculés sans les Néandertaliens.
En utilisant ces estimations pour les taux de mutations, on peut déterminer la date du MRCA de tous les hommes modernes à environ 157.000 ans, et la date de la divergence entre tous les hommes non africains à environ 78.300 ans (entre 62.400 et 94.900 ans). L'émergence de l'haplogroupe Q, sensé dater le peuplement de l'Australie, est estimé à 42.000 ans et l'émergence de l'haplogroupe U5, sensé dater le peuplement de l'Europe, est estimé à 29.600 ans.
Conclusion
Les auteurs ont reconstruit complètement 3 séquences mitochondriales anciennes et presque complètement 6 séquences mitochondriales anciennes, sur un intervalle de 40.000 ans. Toutes les séquences européennes paléolithiques ou mésolithiques sont de l'haplogroupe U. Deux des trois individus de Dolni Vestonice, appartenant à la culture gravettienne, ont la même séquence mitochondriale indiquant une relation maternelle proche. Cette séquence est proche de la base de l'haplogroupe U5. Les individus de Dolni Vestonice ont été datés de 31.500 ans, une date proche de l'âge de l'haplogroupe U5 estimé à 29.600 ans. Les résultats en Europe montre une continuité génétique au paléolithique entre les âges pré-glaciaire et post-glaciaire, jusqu'au début du néolithique. Les estimations des taux de mutations donnent des valeurs 1,6 fois plus élevées que les estimations calibrées par les restes fossiles. Ces estimations ont conduit à une estimation de la divergence entre les africains sub-sahariens et les non africains, inférieure à 95.000 ans, ce qui contredit les résultats obtenus à partir des comparaisons des génomes entre parents et enfants

