Anna Olivieri vient de publier un nouveau papier sur l'analyse de deux haplogroupes mitochondriaux rares: I et W: Mitogenomes from Two Uncommon Haplogroups Mark Late Glacial/Postglacial Expansions from the Near East and Neolithic Dispersals within Europe.
Une question fondamentale concerne les quantités relatives de flux génétique issues de migrations paléolithiques et de migrations néolithiques chez l'européen moderne. Les migrations paléolithiques concernent à la fois la première migration en Europe et la repopulation de l'Europe à partir des refuges glaciaires après la dernière glaciation. Les premières analyses indiquaient le rôle majeur du néolithique, alors que les études plus récentes sur l'ADN mitochondrial montrent une part plus importante du mésolithique dans le génôme européen. Ainsi Maria Pala, dans une étude récente, suppose que les haplogroupes mitochondriaux J et T se sont répandus en Europe avant le néolithique. Cependant de nombreuses questions restent sans réponse, mais le développement des tests d'ADN ancien peut aider à résoudre ces problèmes.
Les premières études d'ADN ancien mitochondrial semblent montrer une discontinuité entre les chasseurs-cueilleurs principalement de l'haplogroupe U et les premiers fermiers néolithiques. Ces résultats suggèrent que le néolithique s'est diffusé principalement par migrations en provenance du Proche-Orient. De plus la différence entre les haplogroupes des premiers fermiers et les haplogroupes des européens modernes supposent des événements migratoires post-néolithiques importants. La situation est donc plus compliquée que le laissaient entendre les premiers scénarios.
Les progrès récent en séquençage de l'ADN mitochondrial a vu une augmentation importante des génomes mitochondriaux complets disponibles dans les bases de données publiques. L'arbre phylogénétique devient ainsi beaucoup plus précis et donne de nombreuses informations. L'étude présentée ici se focalise sur deux haplogroupes ouest-eurasiens: I et W qui sont largement distribués sur tout le continent européen, le Proche-Orient et l'Asie de l'ouest, mais à basse fréquence. Ces deux haplogroupes sont issus directement des haplogroupes N1 et N2 proche de la branche N qui est la première branche non africaine, qui est entrée d'abord en Asie du sud-ouest il y a 60.000 ans, puis en Europe il y a 45.000 ans.
La phylogénie mitochondriale actuelle n'est pas également répartie, mais favorise largement l'Europe Centrale et du Nord. Ceci affecte notamment les haplogroupes I et W dont les composantes sud-européennes et du Proche-Orient sont sous représentées. Les objectifs de cette analyses sont ainsi de faire le bilan de toutes les séquences disponibles (192 I, 4 N1a1b1 et 223 W), dont 58 nouvelles séquences complètes principalement d'Europe du sud et du Proche-Orient, et enfin rechercher des corrélations entre des sous-clades et des événements démographiques. Ces résultats montrent que les haplogroupes I, N1a1b1 et W sont probablement originaire du Proche-Orient durant le dernier maximum glaciaire, et comme les haplogroupes J et T, ils sont impliqués dans des expansions post-glaciales à partir du Proche-Orient. Ces données contribuent donc à mieux définir le repeuplement de l'Europe entre le dernier maximum glaciaire et avant le début du néolithique.
58 nouveaux échantillons ont été complètement séquencés: 31 W, 26 I ET 1 N1b1A1, et ajoutés à 361 séquences déjà connues pour obtenir un total de 419 séquences. L'arbre phylogénétique a été construit à partir de l'ensemble de ces séquences, et les datations des différentes sous-clades ont été estimées à partir du taux de mutation de Soares. L'haplogroupes N1a1b1 est extrêment rare et représenté par seulement 4 séquences dont 1 de Sibérie et les 3 autres du Proche-Orient.
L'haplogroupe I inclue 192 séquences complètes. N1a1b est daté d'environ 29.000 ans, alors que I est daté entre 20.100 et 21.100 ans, durant le dernier maximum glaciaire. Il donne ensuite naissance à sept branches majeures de I1 à I7.

L'haplogroupe I1 est le plus fréquent des sous-clades de I. Il émerge il y a environ 16.000 ans. On le retrouve principalement en Europe et au Proche-Orient, mais aussi en Afrique du Nord et dans le Caucase. Il se sous-divise en 3 sous-clades: I1a, I1b et I1c qui émergent il y a 10.000 à 13.000 ans. I1a se retrouve principalement en Europe:
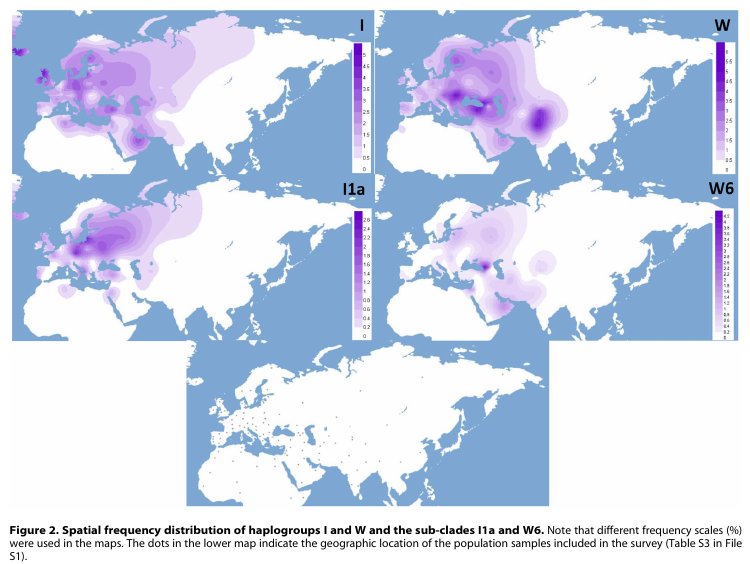
Les haplogroupes I2 et I3 sont issus d'un même noeud et ont émergé entre 6.800 et 10.600 ans. Ces haplogroupes se retrouvent principalement en Europe, mais aussi au Proche-Orient. Les sous-clades I2a et I2b se retrouvent seulement en Europe et sont datées de 4700 et 1700 ans. L'haplgroupe I4 est daté de 15.100 ans et se divise ensuite en I4a et I4b datés de 6400 et 8400 ans. On les retrouve en Europe, au Proche-Orient et dans le Caucase. L'haplogroupe I5 est le second sous-groupe de I en fréquence et se retrouve en Europe et au Proche-Orient. Il est daté de 18.500 ans. I5a1 se retrouve seulement en Europe et est daté de 9.200 ans. Au contraire l'haplogroupe I6 est très rare et se retrouve principalement au Proche-Orient. Enfin l'haplogroupe I7 se retrouve seulement dans deux échantillons. Il est daté de 9100 ans.
L'haplogroupe W inclue 223 séquences complètes. Il émerge il y a environ 17.000 ans. Il s'étend du nord de l'Inde à l'Europe de l'ouest. Il se divise en 6 sous-clades: W1, W3 à W7. L'haplogroupe W1 est le plus fréquent. Il est daté de 10.400 ans et diverge en 6 sous-clades de W1a à W1c et W1e à W1g. W1a et W1b sont datés respectivement de 1600 et 2800 ans. On les retrouve en Finlande et en Europe du Nord. W1c est daté de 8000 ans et se retrouve en Europe, au Proche Orient, dans le Caucase et en Inde. L'haplogroupe W3 est daté de 15.000 ans.et s'étend de l'Europe jusqu'à l'Inde. Il se divise en 2 sous-clades: W3a et W3b datées de 12.000 et 10.000 ans. Les sous-clades W4, W5 et W6 datent approxiamtivement de la même période entre 12.200 et 12.600 ans, mais sont distribuées différemment. W4 s'étend en Europe, au Proche-Orient, en Mongolie et en Inde. W5 se trouve principalement en Europe et au Maghreb. W5a et W5b datés respectivement de 8400 et 9300 se retrouvent exclusivement en Europe. L'haplogroupe W6 est daté de 12.600 ans et s'étend en Europe de l'est et au Proche-Orient. Son pic de fréquence se situe au Caucase. Enfin l'haplogroupe W7 daté de 10.000 ans environ est représenté par seulement 4 échantillons.
Les figures de Bayesian Skyline Plot montrent deux expansions principales pour l'haplogroupe N1a1b: la première se situe entre 11.000 et 13.000 ans, et la seconde à partir de 7000 ans, et une expansion unique entre 13.000 et 7.000 ans pour W:

Discussion
Il est probable que les haplogroupes I et W ont émergé au Proche-Orient durant le dernier maximum glaciaire. Ils semblent très similaires aux 2 haplogroupes précédemment étudiés: J et T. Ces deux haplogroupes ont récemment été étudiés. Il a été proposé que leur expansion et leur entrée en Europe à partir du Proche-Orient, s'est déroulé après le dernier maximum glaciaire, mais avant le néolithique. Ce scénario pourrait être identique pour les haplogroupes I et W, notamment signalé par les sous-clades I1, I2'3, I5, W3, W4 et W5.
Les auteurs de cette étude proposent donc que la majorité des haplogroupes européens sont arrivés en Europe avant l'arrivée des premiers fermiers. Pour ma part, je suis très sceptique sur les conclusions de cette étude. Je ne vois pas que les données indiquent une expansion en Europe pré-néolithique. Je penche davantage pour une première expansion de ces haplogroupes limitée au Proche-Orient, suivie d'une seconde expansion avec les premiers fermiers du néolithique. C'est selon moi, le moment où ces haplogroupes sont arrivés en Europe. C'est en tout cas ce que montre les résultats actuels des tests d'ADN ancien. Cela doit être également le cas pour les haplogroupes J et T.

