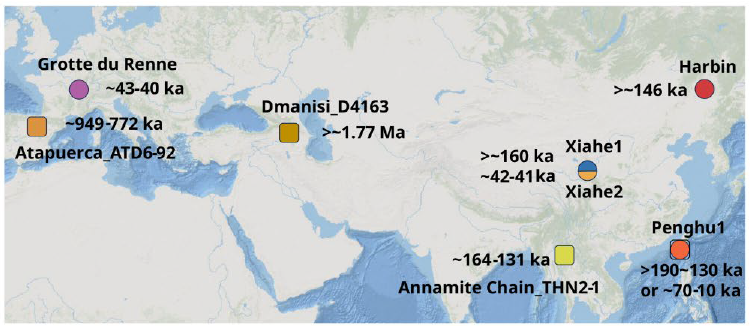
Les Dénisoviens sont un groupe d'anciens hominidés d'Asie identifié à partir de l'analyse ADN de fossiles issus de la grotte Denisova située en Sibérie. Les fossiles actuels attribués à l'homme de Denisova représentent quelques dents et quelques fragments d'os. Des analyses protéomiques précédentes ont également permis d'identifier une partie de mandibule et une côte de la grotte Baishiya située au Tibet, une mandibule issue du canal Penghu à Taïwan et une molaire issue de la grotte Cobra au Laos comme appartenant à des Dénisoviens. Ainsi, malgré l’identification des Dénisoviens comme une lignée humaine archaïque distincte par rapport aux Néandertaliens et aux humains modernes, nous ne savons toujours pas à quoi ils ressemblaient. Cependant, certains fossiles (Xujiayao, Harbin), avec des crânes presque complets et des caractéristiques morphologiques informatives, ont été suggérés comme de possibles Dénisoviens, compte tenu de leur âge, de leur géographie et de leurs caractéristiques archaïques.
Le fossile de Harbin est un crâne quasiment complet situé dans la province de Heilongjiang en Chine. Sa datation a été estimée supérieure à 146.000 ans à partir de mesures de datation uranium-thorium, alors que la stratigraphie de la couche dans laquelle ce fossile a été retrouvé, suggère une date comprise entre 138.000 et 309.000 ans. Dans un papier précédemment publié, ce fossile a été attribué à une nouvelle espèce: Homo longi, littéralement l'homme dragon, à partir de sa morphologie.
Qiaomei Fu et ses collègues viennent de publier un papier intitulé: The proteome of the late Middle Pleistocene Harbin individual. Ils ont analysé le protéome à partir des os du rocher du crâne de Harbin.
Les auteurs ont obtenu 95 anciennes protéines appartenant à l'homme de Harbin. Ces résultats montrent une connexion claire avec les Dénisoviens. Ainsi une allèle dérivée chez l'homme de Harbin, a auparavant été détectée chez les individus de la grotte Baishiya au Tibet et du canal Penghu à Taïwan. Les auteurs ont ensuite tracé un arbre phylogénétique afin de comparer le protéome de l'homme de Harbin avec celui de différentes espèces:
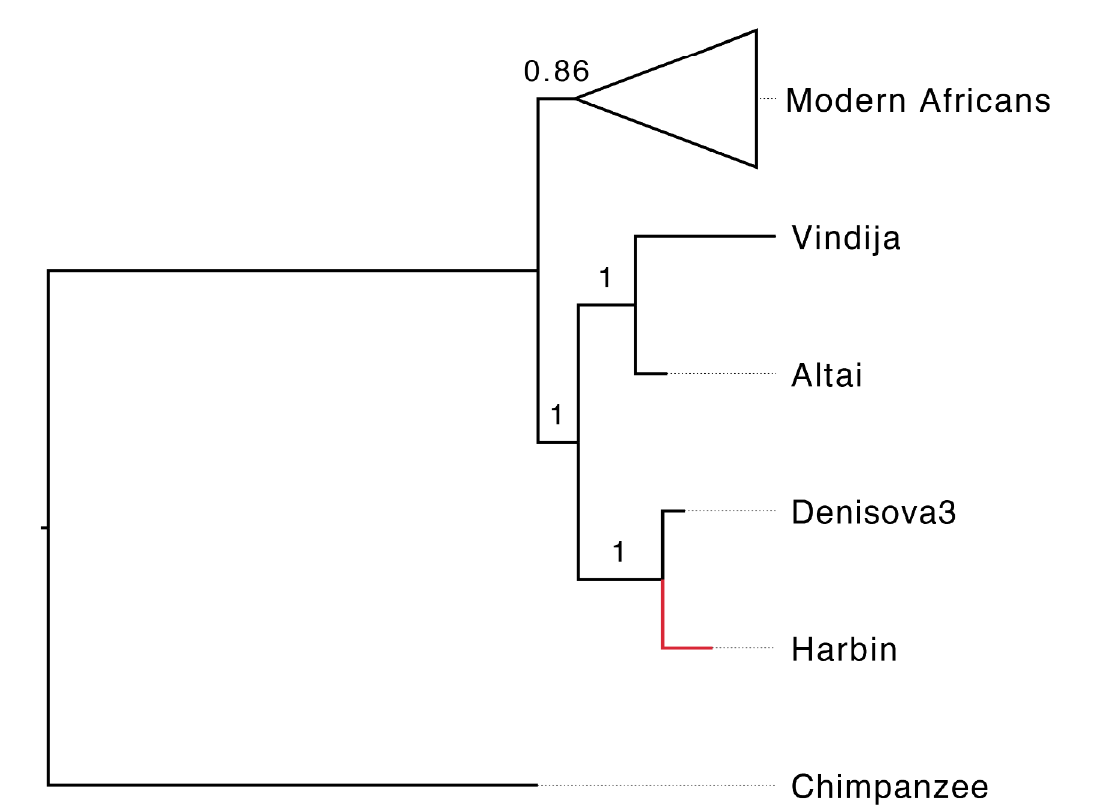
Les résultats montrent clairement le rattachement de l'homme de Harbin avec les Dénisoviens et son éloignement des hommes de Néandertal (Vindija, Altaï) et des hommes modernes. Ces résultats suggèrent que l'homme de Denisova était largement répandu en Asie au Pléistocène. De plus il permet de savoir à quoi ressemblait morphologiquement l'homme de Denisova.
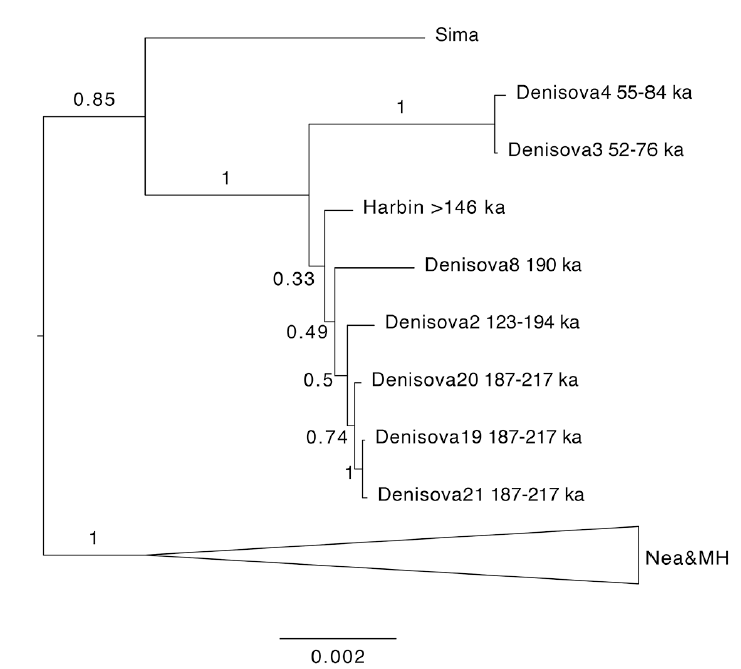
Les auteurs ont également publié un papier intitulé: Denisovan mitochondrial DNA from dental calculus of the >146,000-year-old Harbin cranium. Ils ont essayé d'analyser le génome de l'homme de Harbin. Malheureusement, ils ont échoué à récupérer de l'ADN autosomal à partir de l'os du rocher ou à partir d'une dent. Ils ont néanmoins réussi à obtenir le génome mitochondrial de l'homme de Harbin à partir du tartre dentaire.
Les résultats montrent que le génome obtenu contient des allèles dérivés spécifiques aux Dénisoviens. Les auteurs ont ainsi reconstitué un arbre phylogénétique afin de comparer le génome mitochondrial obtenu avec celui d'autres Dénisoviens, d'hommes de Néandertal, d'hommes modernes ainsi que de l'homme de Sima de los Huesos. Le résultat obtenu montre que l'homme de Harbin se situe dans la variabilité des Dénisoviens:
mardi 24 juin 2025
L'homme de Harbin en Chine est un Dénisovien
mardi 24 juin 2025. Protéome ancien
samedi 21 juin 2025
ADN ancien en Colombie entre 6000 et 500 ans
samedi 21 juin 2025. ADN ancien
Les études précédentes de paléo-génétique ont montré que les populations Amérindiennes en dehors de la zone arctique, dérivent d'un mélange génétique entre une population est asiatique et une population sibérienne qui a eu lieu quelque part dans le nord est de l'Asie il y a plus de 20.000 ans. Il y […]
dimanche 15 juin 2025
Transition entre le Néolithique et l'Âge du Bronze à Mas d’en Boixos en Catalogne
dimanche 15 juin 2025. ADN ancien
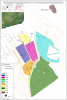
Mas d’en Boixos est le site archéologique le plus significatif de la région de Penedès en Catalogne située entre Barcelone et Tarragone. Les fouilles débutées en 1997 ont livré 450 structures datées entre le début du Néolithique et la fin de l'Âge du Fer soit entre 5500 et 200 av. JC. Ce site est […]
samedi 3 mai 2025
Continuité génétique dans la culture des Pueblos au Nouveau Mexique
samedi 3 mai 2025. ADN ancien

Les anciens pueblos créés par la culture ancestrale des Pueblos, un terme indiquant leur relation ancestrale avec les tribus amérindiennes contemporaines des Pueblos, comptent parmi les cultures archéologiques les plus reconnaissables d'Amérique du Nord. De nombreux pueblos anciens sont devenus le […]
vendredi 25 avril 2025
Anciens génomes des communautés Phéniciennes et Puniques
vendredi 25 avril 2025. ADN ancien
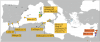
La culture phénicienne a émergé dans des cités états de l'Âge du Bronze du Levant. Au début du premier millénaire av. JC., les phéniciens ont établi un vaste réseau commercial le long des côtes méditerranéennes jusque sur les côtes sud-ouest de la péninsule Ibérique, diffusant leur culture, leur […]
lundi 14 avril 2025
La mandibule d'un homme de Denisova à Taïwan
lundi 14 avril 2025. Protéome ancien
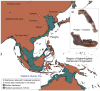
Les Dénisoviens ont été reconnus comme un groupe humain distinct des hommes modernes et des hommes de Néandertal par l'analyse ADN de restes osseux et dentaires issus de la grotte de Denisova située dans l’Altaï au sud de la Sibérie. Leur génome nucléaire a montré qu'ils formaient un groupe frère […]
lundi 7 avril 2025
Génomes anciens du Sahara vert
lundi 7 avril 2025. ADN ancien
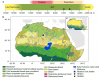
Après le dernier maximum glaciaire, une transformation climatique au Sahara a conduit à une période Africaine humide qui a été maximale entre 11.000 et 5000 ans. A cette époque le Sahara était une savane dans laquelle le paysage alternait entre couverture arborée variable, lacs permanents et système […]
dimanche 30 mars 2025
Histoire génétique de la région Rhin-Meuse
dimanche 30 mars 2025. ADN ancien
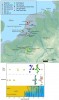
La région Rhin-Meuse s'étend au Pays-Bas, en Belgique et dans le nord-ouest de l'Allemagne. Elle est caractérisée par un environnement riche en eau. Vers 5500 av. JC. le sud de cette région voit l'arrivée de fermiers colons appartenant à la culture rubanée qui se sont installés dans des zones […]
lundi 17 mars 2025
Génomes anciens en Afrique du nord
lundi 17 mars 2025. ADN ancien

Sur la côte Méditerranéenne de l'Europe, la transition néolithique se caractérise par la migration de populations venues d'Anatolie qui ont atteint la péninsule Ibérique il y a 7500 ans. Ces populations ont absorbé jusqu'à 30% d'ascendance des chasseurs-cueilleurs de l'ouest. La culture est alors […]
mardi 4 mars 2025
Diffusion des langues celtiques
mardi 4 mars 2025. ADN ancien

Le paysage linguistique de l'Europe actuel est dominé par les langues Romanes, Germaniques et Slaves qui se sont diffusées relativement récemment durant l'Âge du Fer et après l'émergence de l'empire Romain et des migrations barbares. Cependant avant ces événements, les langues celtiques dominaient […]
mardi 25 février 2025
Anciens génomes de nourrissons sur le site de Lepenski Vir en Serbie
mardi 25 février 2025. ADN ancien

Un des rares sites archéologiques qui témoigne des interactions entre chasseurs-cueilleurs et fermiers est localisé à Lepenski Vir proche de la frontière entre la Serbie et la Roumanie. Les fouilles dans les gorges du Danube ont révélé des habitats et des sépultures fournissant plus de 500 restes […]
lundi 10 février 2025
Transition entre la fin du Néolithique et le début de l'Âge du Cuivre dans le bassin des Carpates
lundi 10 février 2025. ADN ancien

La géographie du bassin des Carpates en fait un carrefour entre l'Europe du sud-est et l'Europe centrale. Les vallées fertiles du Danube et de ses affluents offrent des conditions optimales pour l'installation des habitats préhistoriques et pour faciliter leurs réseaux de communication et d'échange. […]
mardi 21 janvier 2025
Matrilocalité dans la Grande-Bretagne de l'âge du fer
mardi 21 janvier 2025. ADN ancien

Une société matrilocale dans laquelle les époux viennent habiter auprès des parents de leur femme est relativement rare, alors que les sociétés patrilocales sont beaucoup plus nombreuses. De plus dans la plupart des anciennes sociétés préhistoriques d'Europe avec suffisamment de données […]
mercredi 8 janvier 2025
Relations familiales dans des sépultures de l'élite Moche du Pérou vers l'an 500
mercredi 8 janvier 2025. ADN ancien

La culture archéologique Moche s'est étendue dans neuf vallées le long de la côte nord du Pérou entre l'an 300 et l'an 950 de notre ère. Elle a laissé des complexes urbains incluant des temples monumentaux en forme de pyramide (huacas), des réseaux d'irrigation, et des œuvres artistiques élaborées […]
mardi 17 décembre 2024
Les plus anciens génomes d'homme moderne d'Europe
mardi 17 décembre 2024. ADN ancien

A ce jour, seulement cinq sites archéologiques ont produit un génome d'homme moderne plus ancien que 40.000 ans: Tianyuan en Asie de l'est daté d'environ 40.000 ans, Ust'Ishim en Sibérie daté d'environ 44.000 ans, Bacho Kiro en Bulgarie daté d'environ 44.000 ans, Oase en Roumanie daté d'environ […]
lundi 9 décembre 2024
La paléo-génétique supporte une ancienne divergence entre les langues Indo-européennes de l'est et de l'ouest de la Méditerranée
lundi 9 décembre 2024. ADN ancien

Depuis 5000 ans, des migrations humaines significatives ont restructuré le profil génétique des populations d'Eurasie. Différentes vagues d'ascendance de la steppe associées aux pasteurs de la culture Yamnaya se sont diffusées sur de longues distances. Ces migrations ont également joué un rôle clef […]
mardi 3 décembre 2024
Lignages féminins et relations familiales à Çatalhöyük
mardi 3 décembre 2024. ADN ancien

Un long débat existe au sujet du culte de la déesse mère dans les premières sociétés Néolithiques. Cette théorie s'inspire des nombreuses figurines féminines retrouvées en Anatolie et dans la région Égéenne interprétées comme des déesses symbolisant la fertilité ou représentatives de sociétés […]
lundi 18 novembre 2024
Analyse bio-moléculaire des restes humains épi-gravettien du site de Riparo Tagliente dans le nord de l'Italie
lundi 18 novembre 2024. ADN ancien

L'abri sous roche de Riparo Tagliente est situé dans la province de Vérone au nord-est de l'Italie. Il est associé à une abondante collection d'ossements animaux et quelques os humains. Il a été découvert en 1958 avant d'être fouillé entre 1962 et 1964 par le musée d'histoire naturelle de Vérone, […]
samedi 2 novembre 2024
L'émergence et la transformation des pasteurs de l'Âge du Bronze dans le Caucase
samedi 2 novembre 2024. ADN ancien

La région du Caucase et son voisinage correspondent à une interface entre l'Europe et l'Asie. Au milieu de l'Holocène, les hautes montagnes du Caucase ont fonctionné comme une barrière semi-perméable à travers laquelle les idées, les technologies, les langues et les peuples ont bougé. Avec son […]
lundi 21 octobre 2024
Génome d'un ancien énéolithique issu du cimetière de Nalchik situé au nord du Caucase
lundi 21 octobre 2024. ADN ancien
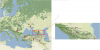
A la fin du septième et au début du sixième millénaire av. JC., le processus de néolithisation en cours en Asie de l'ouest a atteint les plaines situées au sud du Caucase. Ainsi, les anciens sites de Shulaveri, Shomutepe et Aratashen sont situés dans les vallées Kura et Araxes. Il n'y a pas de […]
« billets précédents - page 1 de 33

